(通讯员 董科)4月26日,登录入口曹罡课题组与李国亮课题组在国际著名期刊Nature Genetics联合发表题为“Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture”的研究论文。该论文介绍了一种新染色体构象捕获技术(DLO Hi-C),为解析基因组三维结构提供了一种新型高效、经济的研究方法。登录入口博士生林达与信息学院博士生洪萍为论文的共同第一作者,曹罡教授与信息学院李国亮教授为论文的共同通讯作者。
基因编码区在基因组序列中只占极少部分。近年来研究表明其余“基因组垃圾序列”在基因转录调控中发挥着重要的调控功能。对染色质构象和三维基因组学的研究可以将基因组上原本分散的远距离调控元件与其具体调控区域更好的关联起来,对理解基因的转录调控、增强子与启动子的相互作用、疾病易感位点、DNA损伤修复、基因组结构变异和表观遗传有着重要的意义。
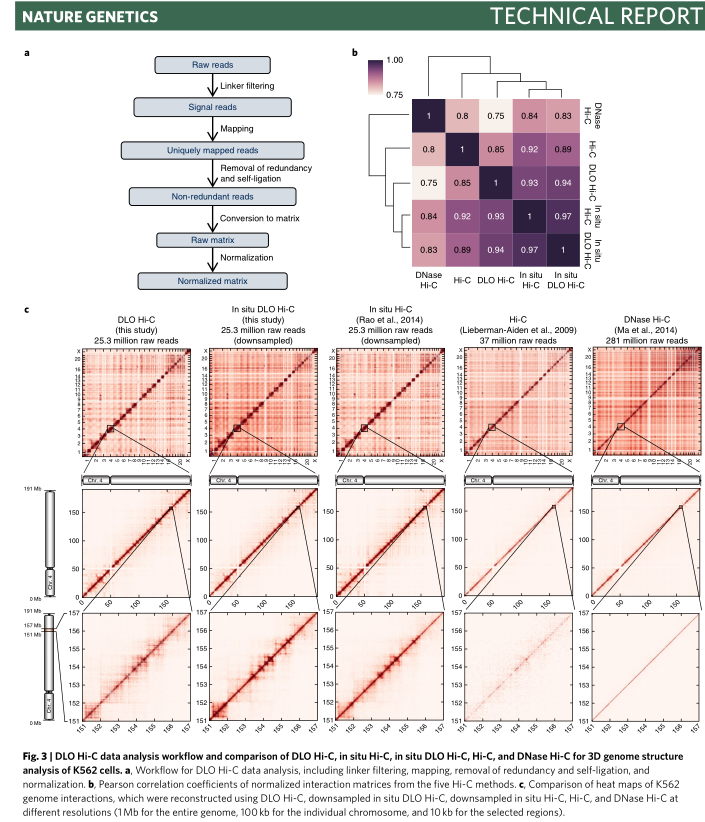
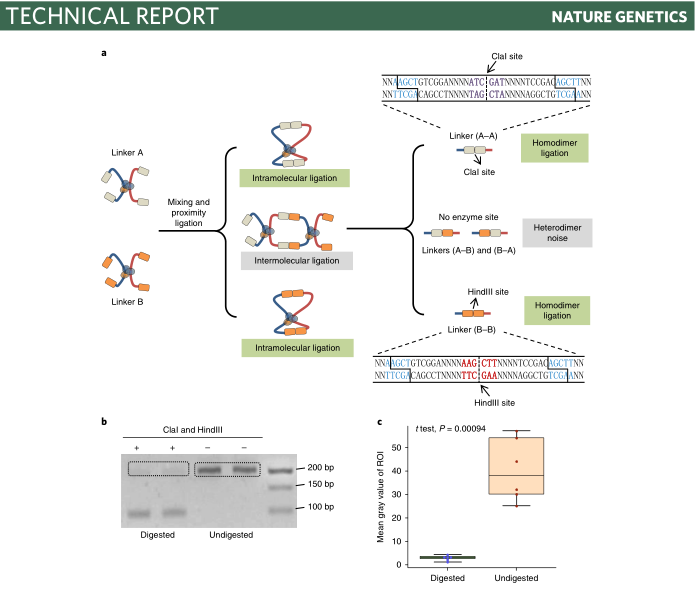
Hi-C技术是三维基因组学的主要研究方法,但存在实验成本高、数据噪音大、实验过程繁琐等缺点。为解决这些问题,该论文提供了一种新的DLO Hi-C染色体构象捕获技术,采用同时酶切酶连的方式,将DNA接头连接在染色体内切酶切口末端上,然后进行邻近酶连,最后将用MmeI内切酶酶切消化,回收固定大小互作DNA片段,从而大大地降低了背景数据噪音,获取质量高于传统Hi-C的数据。DLO Hi-C技术还可以用于染色体易位位点筛选。
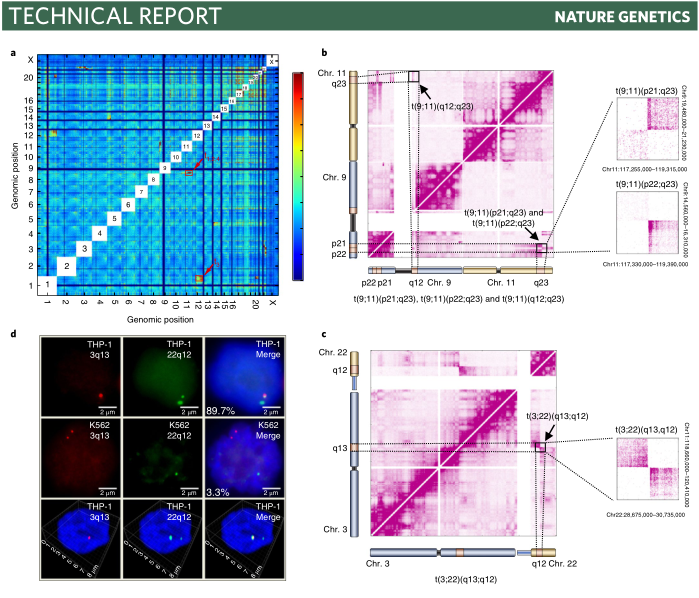
DLO Hi-C技术使全基因组染色体构象捕获实验的成本大大的降低,同时简化了实验步骤,使得实验成功率显著提高,对辅助基因组组装、解析基因组远程调控元件的功能、理解疾病易感位点、以及检测染色体结构变异有着重要的意义。Nature Genetics同期发表专门评论性文章,对此项研究工作给予了较高评价“Lin et al. introduce an elegant dual-linkerstrategy that allows for noise filtering and early quality control.”
文章链接:https://www.nature.com/articles/s41588-018-0111-2
同期评论文章链接:https://www.nature.com/articles/s41588-018-0112-1
审核人:曹罡

